Más de 700 mil muertes anuales se presentan cada año en el mundo debido a infecciones por bacterias resistentes a los antimicrobianos, lo que se ha convertido en un serio problema de salud pública mundial, que podría ocasionar 10 millones de muertes en los próximos 25 años y dejar pérdidas económicas que superarían los 100 billones de dólares para 2050.
(Organización Mundial para las Salud marzo 2021).
En el laboratorio clínico a través del área de microbiología clínica, somos los primeros en identificar a estas cepas multirresistente e interpretar sus perfiles de resistencia, esto nos convierte en uno de los ejes centrales para el
diagnóstico y manejo oportuno.
Cepas multirresistentes
Hace algunos años se tenía presente que la resistencia bacteriana podría clasificarse en relación a la forma en que se adquiere la infección; de ahí podemos mencionar dos tipos: cepas que provenían de infecciones comunitarias, con perfiles de resistencia bajos; y las nosocomiales, las cuales presentaban características y mecanismos de resistencia antimicrobiana(RAM) más pronunciados y difíciles de tratar; en la actualidad existe diferentes evidencias que esto ha quedado desestimado, ya que hoy en día la gran mayoría de cepas que provienen de infecciones comunitarias presenta altos perfiles de resistencia ,un ejemplo muy evidente son las bacterias perteneciente a la familia Enterobacteriales, entre ellas Escherichia coli, que es uno de los principales patógenos asociados a ITU(Infecciones tracto urinario) , la cual no presenta resistencia intrínseca, pero en los últimos años demuestra altos niveles resistencia adquirida como de tipo BLEEE(Betalactamasa de espectro extendido) o incluso a antibióticos de amplio espectro como los carbapenémicos.
En el 2023 la Organización Mundial para la Salud (OMS) ha publicado una lista de microrganismos los cuales se han denominado en base a prioridad de resistencia y clasificados en 3 categorías; estos microrganismos son:
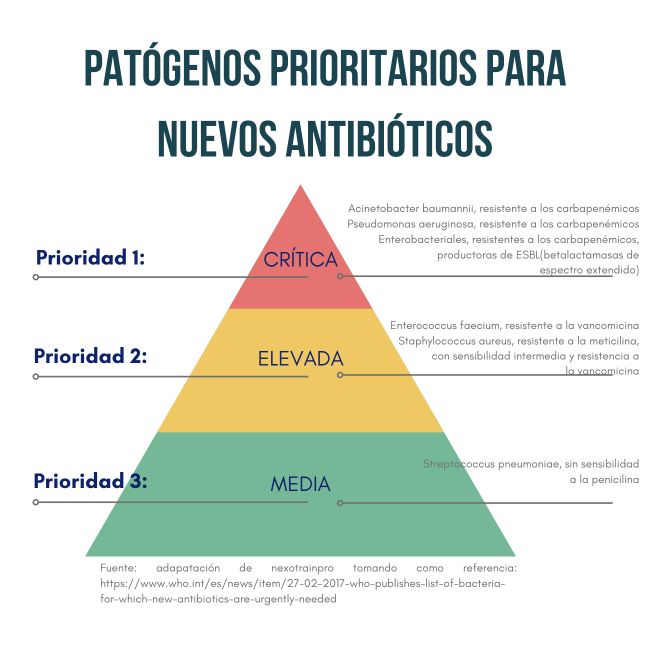
Retos en el diagnóstico en el laboratorio de microbiología
En la actualidad existen diferentes métodos automatizados utilizados para la identificación de las bacterias causantes de infecciones, realizando pruebas de susceptibilidad y además algunos de estos equipos brindan informes de los mecanismos de resistencia que estas presentan; tecnologías como VITEK, MicroScan y MALDI TOF, por mencionar algunas. Estas tecnologías proporcionan una optimización y rapidez en los resultados, personalmente considero que estos recursos son de vital importancia, pero no reemplaza la realización de pruebas bioquímicas manuales convencionales o la identificación de las características microscópicas y culturales(macroscópicas de las colonias) , las cuales brindan detalles muy valiosos en la identificación; incluso de forma preliminar evidencia de resistencia ;como por ejemplo la hirpermucosidad de algunas colonias, entre otros aspectos.
Para la identificación de estas cepas bacterianas resistentes es importante tomar en cuenta los siguientes aspectos:
- Tipos de muestras clínicas y bacterias patógenas: la identificación de las especies bacterianas presentes en las muestras clínicas es una disciplina aplicada de la microbiología de gran significación práctica. Diferenciar si estamos ante un
microorganismo patógeno, uno potencialmente patógeno o un contaminante y deducir los posibles mecanismos de resistencia del microorganismo, conduce a tratar la enfermedad infecciosa con la mayor eficiencia posible, con antimicrobianos u otras terapias antiinfecciosas, y todo procurando el menor coste posible
- Evaluación de las características culturales y microscópicas de las cepas: Un aspecto muy relevante a tomar en cuenta es que las bacterias han sufrido cambios fenotípicos y genéticos , los cuales se muestran hasta en el tamaño, color, forma y
consistencia de las colonias, ejemplos de estos son: Escherichia coli una bacteria que presentaba características muy marcadas en medios selectivos y diferenciales como Agar Mac Conkey , en el cual se observaban colonias rosadas por la fermentación rápida de la lactosa, (halo rosado) resecas, medianas y pequeñas.En la actualidad esto se puede apreciar diferente manera, ya que algunas de estas cepas son lactosa negativo en el y presentan consistencia mocosa agar antes mencionado.Otro aspecto muy importante es no olvidar la realización de la coloración de Gram, la cual sigue siendo el estándar de oro para observación de características microscópicas, así como la presencia de PMN (polimorfonucleares) a partir de las muestras (su realización e interpretación depende del tipo de muestra). - Realización de pruebas bioquímicas manuales de género y especie: Es muy importante que el área de bacteriología se cuente siempre con estas pruebas, ya que estos nos ayudan a confirmar los reportes establecidos por los equipos. Hay pruebas que son indispensables para definir géneros bacterianos; como las pruebas de: catalasa para cocos Gram positivos y la oxidasa para bacilos Gram negativos.
- Una correcta interpretación de los reportes y perfiles de resistencia: Este es uno de los aspectos más relevantes para el control y manejo de estas cepas en los centros hospitalarios y laboratorios clínicos. Hay que recordar algo, y es que el equipo por más innovador que sea, no puede superar nuestros conocimientos. Muchos al no tomarle importancia a este punto, brindan todos los reportes que el equipo proporciona sin antes hacer una interpretación correcta, a continuación, citare algunos ejemplos. En la identificación de Staphylococcus aureus con resistencia de tipo MLSB (Macrólidos, Lincosamidas y Estreptogaminas B) los equipos como VITEK realizan el D test brindando un resultado como positivo o negativo , pero la interpretación de este tipo de resistencia es muy importante, ya que el equipo cuando este test es positivo brinda el informe del perfil de antibióticos: Eritromicina Resistente y Clindamicina Sensible, en este caso debe reportarse el antibiótico de Clindamicina como resistente ya que un D test positivo nos indica que hay un mecanismo resistencia de tipo inducible, lo cual ha exponer este antibiótico a la bacteria, esta emitirá el mecanismo de resistencia, y de forma invivo provocar un posible fracaso terapéutico, además es conveniente que este test también se pueda confirmarse de forma manual en el laboratorio. Otro de los ejemplos evidentes es cuando el equipo informa BLEE + y que el reporte de antibióticos emite un resultado sensible para cefalosporinas de 3° o 4° generación, las cual deben ser cambiadas ha resistente, ya que no habría una concordancia debido al espectro de acción que abarca estas betalactamasas.
- Actualización Continua: Una de las áreas que constantemente se actualizan es la de microbiología clínica, ya que exiten diferentes guías internacionales que estandarizan estos procesos, por ejemplo, los documentos CLSI-Instituto de
Estándares Clínicos y de Laboratorio (tablas M-100) o EUCAST(Comité Europeo de Pruebas de Susceptibilidad a los Antimicrobianos) estos establecen los puntos de corte de los antibióticos y su interpretación correcta; estas guías se actualizan cada año basándose en estudios epidemiológicos e investigaciones. Comparto una de las nuevas actualizaciones desde el año 2023, y es la nueva clasificación para el uso de antibióticos en las pruebas de susceptibilidad, que anteriormente era asignado por letras y en la reciente publicaciones se ha considerado agruparlos por niveles en base a la resistencia y prioridad de manejo Fuente:http://antimicrobianos.com.ar/wpcontent/uploads/2023/11/NOVEDADES-
CLSI-2023.pdf. - Comunicación asertiva: Por último, crear canales de comunicación constante con los médicos infectólogos, epidemiólogos, además del personal de salud, brindado y compartiendo información, ya que de esta manera se establece datos concretos, a partir de los cuales se crean los protocolos a seguir para este tipo de bacterias. Nuestra labor no solo debe quedarse dentro del laboratorio, debe pronunciarse y hacerse notar.
Conclusión
Personalmente considero que la bacteriología es una de las áreas con más auge en la actualidad, que como profesionales perdamos el miedo a emitir una opinión sobre este tema, busquemos medios y oportunidades para fortalecer esta área. Otra recomendación es realizar siempre controles de calidad adquiriendo lo necesario, en este caso contar con cepas ATCC, las cuales nos brindan los estándares de calidad en cada proceso en el área de microbiología clínica.
Cada año la OPS conmemora la lucha contra la resistencia antimicrobiana con el enfoque de: One Healt – una sola salud – lo que significa que todo lo que nos rodea es parte de la solución (salud humana, animal y medio ambiente), bajo este concepto desarrollemos un alto compromiso de lucha desde nuestros laboratorios, realizando procesos de calidad
Me despido desando éxitos en sus laborales y compartiendo la siguiente cita: “Sin laboratorios los hombres de ciencia son como soldados sin armas.” Louis Pasteur – Químico Francés
Escrito por: Máster Alexander Alfredo Valle Zometa.
Licenciado e Laboratorio clínico y Máster a en Microbiología Clínica (EDUCA BINESS Granada España) – Docente y Coordinador
de área de Bacteriología.
Lecturas complementarias:
- Identificación de bacterias resistentes a antibióticos carbapenémicos en hospitales de El Salvador: https://camjol.info/index.php/alerta/article/download/7135/7729/58178
- La batalla contra las superbacterias: No más antimicrobianos, no hay ESKAPE: https://www.scielo.org.mx/pdf/tip/v23/1405-888X-tip-23-e20200202.pdf
- Lectura interpretada del antibiograma de cocos Gram positivos: https://www.elsevier.es/es-revista-enfermedades-infecciosas-microbiologia-clinica-28-articulo-lectura-interpretada-del-antibiograma-cocos-S0213005X10000844
- MECANISMOS DE RESISTENCIA EN PSEUDOMONAS AERUGINOSA: ENTENDIENDO A UN PELIGROSO ENEMIGO https://revistas.unal.edu.co/index.php/revfacmed/article/view/43484/44759
- “INTERPRETACIÓN E INFORME DEL ANTIBIOGRAMA. EVALUACIÓN DE LOS SISTEMAS AUTOMATIZADOS EN LOS LABORATORIOS DE MICROBIOLOGÍA” https://sampac.es/images/site/abstracts/SAMPAC%202008_H.pdf


